La première action de cette équipe a été de répondre à une urgence : la gestion des volumes de données importants générés par les analyses réalisées par spectrométrie de masse. Elle a ainsi développé
ePIMS™ (experimental
Proteomics
Information
Management
System *), une infrastructure de gestion des analyses utilisée quotidiennement par les autres membres du laboratoire. Depuis sa mise en service en novembre 2004, ePIMS™ a permis de gérer plus de 10 000 échantillons ayant donné lieu à près de 19 000 analyses sur les spectromètres de masse, représentant plus de 9
To * de données.
D’autres plate-formes protéomiques françaises (Toulouse, Montpellier, Dijon, Paris…) se sont montrées intéressées par cette infrastructure ; plusieurs d’entre elles sont en train de l’implémenter. L’équipe informatique du laboratoire a donc entrepris de valoriser et d’assurer la pérennité d’ePIMS™ par sa diffusion sous la licence libre CeCILL (licence élaborée conjointement par le CEA, le CNRS et l'Inria) et en s’assurant le concours d’une société à laquelle le savoir-faire nécessaire à l’installation et à la maintenance de cette infrastructure sur d’autres plate-formes protéomiques a été transféré.
• Une seconde illustration de cette conception du rôle de l’équipe informatique au sein du laboratoire est le développement du logiciel
IRMa (Interprétation des
Résultats
Mascot). En effet, une partie importante de l’activité du laboratoire EDyP consiste à identifier les protéines présentes dans un échantillon biologique grâce aux spectres de masse MS/MS des peptides (fragments de ces protéines). L’identification fait appel à un moteur de recherche qui compare les spectres théoriques des peptides contenus dans les banques de données protéiques avec les spectres MS/MS obtenus expérimentalement. Malheureusement, de tels moteurs de recherche ne font que proposer une identification potentielle des peptides et donc des protéines ; cette identification doit ensuite être confirmée par une étape de validation qui permet aux experts du Laboratoire de corréler spectres et peptides. IRMa est un outil qui simplifie grandement cette validation grâce à l’application de filtres automatiques tout en assurant la cohérence globale du résultat. Ce logiciel permet à chacun d’évaluer le résultat obtenu et de produire des rapports d’identification ; ces rapports contiennent notamment les spectres MS/MS annotés, éléments désormais indispensables à la publication des résultats dans les meilleures revues scientifiques.
La synergie entre le groupe informatique et le reste de d'équipe permet d’assurer la création d’outils logiciels qui répondent aux besoins exprimés. Cette synergie permet l’émergence de solutions intégrées qui prennent en compte non seulement la diversité des activités en spectrométrie de masse du laboratoire, mais garantissent également l’intégration avec les autres outils développés et les logiciels tiers.
En associant ePIMS™ aux outils d’identification commerciaux et au logiciel de validation IRMa (Figure), il est désormais possible d'enchaîner les étapes de :
- transfert des données depuis les spectromètres (Figure #2)
- extraction des spectres de masse de ces données, (Figure #3)
- recherche d’identification (Figure #4)
- validation du résultat obtenu sans intervention manuelle (Figure #5 et 6).
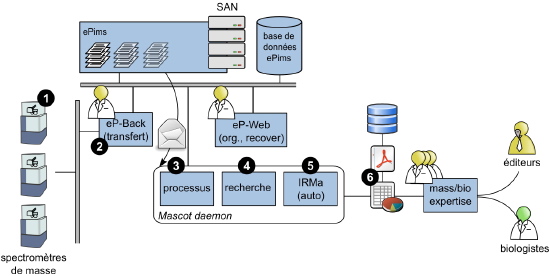
Schéma d’intégration d’ePIMS et d’IRMa dans le processus de production et de validation de la plate-forme de protéomique.
#1 : Acquisition de l’échantillon sur les spectromètres de masse.
L’objectif de cette démarche n’est pas d’exclure l’expertise humaine mais de soulager les scientifiques du laboratoire des tâches à faibles valeurs ajoutées afin de leur donner les moyens de concentrer leurs efforts sur leurs domaines d’expertises.
* 1 To = 1 000 Go.
Une infrastructure de gestion d'analyses, ePIMS™, a été créée en interne et s'appuie sur le réseau de stockage de données mis en place et administré par le
GIPSE de notre laboratoire. La valorisation de ePIMS™ a été entreprise avec l'appui du service de valorisation de la DSV.