Activité de Recherche et Développement
Activité de Recherche et Développement
Stéphanie Bouillot, Technicienne de recherche CEA
Sophie Gerbaud, Ingénieure CEA
Notre objectif est d'utiliser la puissance de l’imagerie automatisée (ou HCS pour High Content Screening) pour la découverte de nouveaux composés actifs dans le contexte cellulaire et pour leur validation et leur caractérisation grâce à un phénotypage cellulaire poussé.
À gauche : IncuCyte ZOOM (Sartorius) permettant le suivi de cellules vivantes avec ou sans marquage (contraste de phase, A488/GFP, Cy3), sur quelques heures à plusieurs jours grâce à un environnement contrôlé en température, CO2 et humidité.
À droite : Microscope CellInsight CX7 confocal LED (Thermo Scientific) pour l’acquisition et l’analyse automatisées d’images de cellules vivantes ou fixées, en brightfield ou fluorescence +/- confocal (7 longueurs d’onde possible), à différents grossissements (4x à 60x).
Le
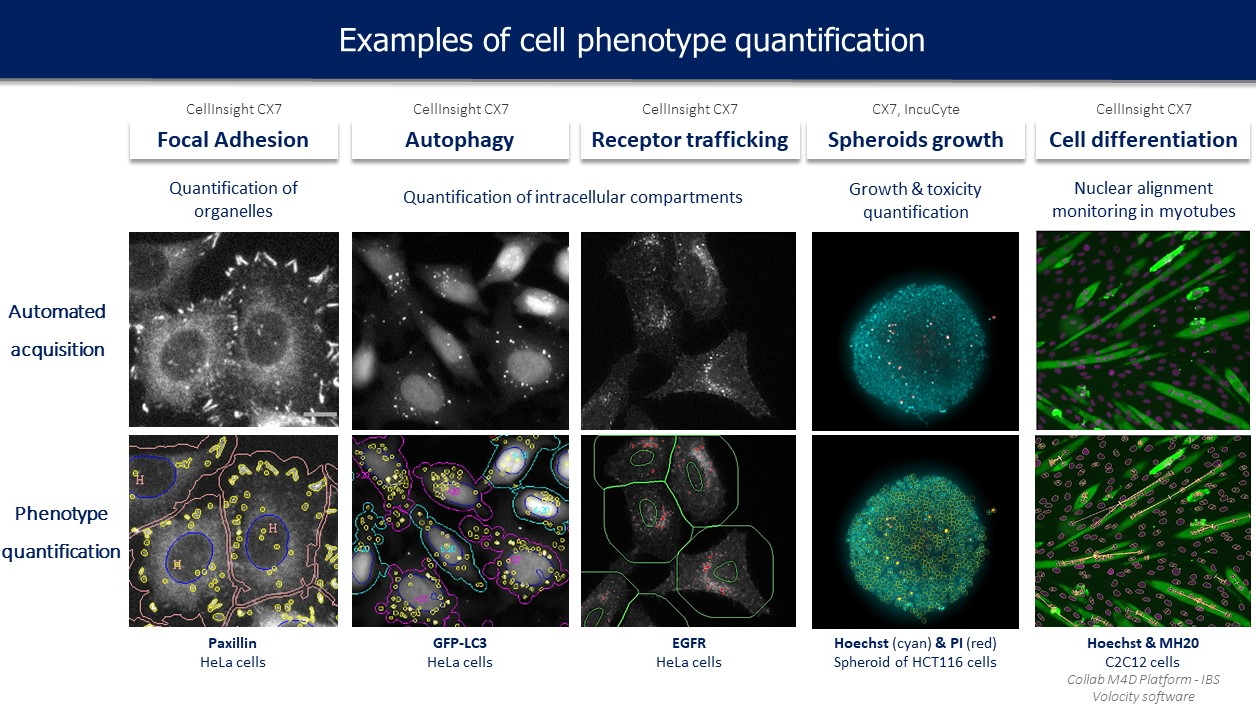
High Content Screening est une technique de criblage par imagerie automatisée sur cellules vivantes ou fixées. Les microscopes utilisés permettent la capture rapide et automatique de grandes quantités d'images de cellules marquées; des logiciels dédiés d'analyse d'images sont utilisés pour extraire automatiquement l'information biologique tels que nombre de cellules/noyaux, analyse des compartiments subcellulaires, analyses morphologiques telles que aire, forme, texture …
En s’appuyant de cette technologie, nous développons une stratégie de phénotypage cellulaire afin d'accélérer la phase de validation et de caractérisation des composés bioactifs sélectionnés par
criblage robotique sur notre plateforme. Différents tests phénotypiques sur cellules ont été miniaturisés en plaques 96 puits et permettent d'investiguer l'effet des petites molécules chimiques sur différents constituants et processus essentiels de la cellule (
Figure 1). Pour chaque test expérimental, des protocoles d'acquisition et d’analyse automatiques d'images sont développés pour une utilisation en routine. La quantité très importante de données générées nécessite le développement et l'intégration de méthodes informatiques de segmentation d'objets (noyaux, cellules, structures subcellulaires…), d'extraction des descripteurs pertinents pour chaque phénotype et de modélisation statistique.
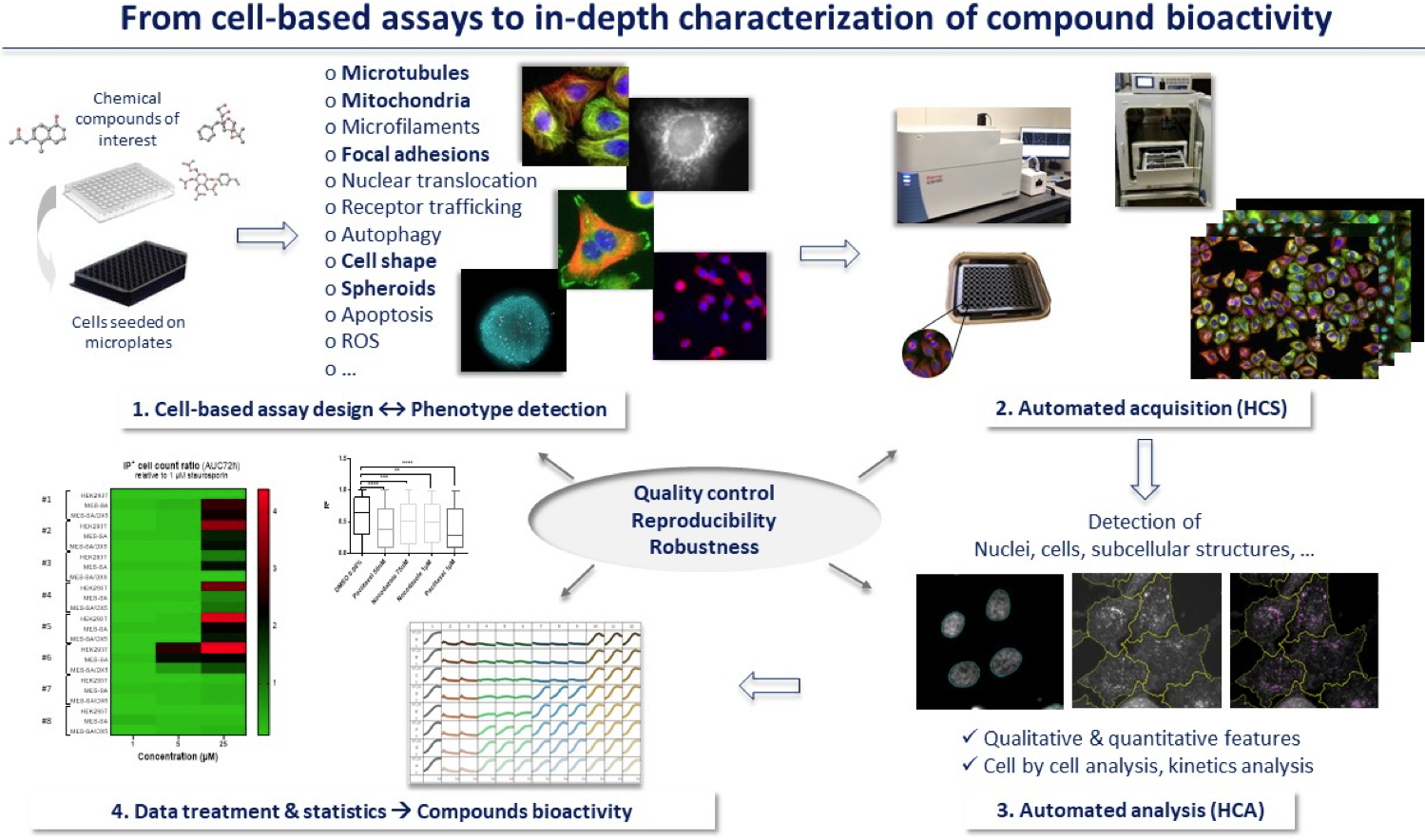 Figure 1 : Stratégie de phénotypage cellulaire.
Figure 1 : Stratégie de phénotypage cellulaire.
Adossé au projet de recherche de l'équipe Gen&Chem, nous appliquons notre stratégie de phénotypage par HCS à la recherche de composés prometteurs pour l'étude et la compréhension des mécanismes de régulation de l'inflammation et de l'immunité innée, et notamment à des composés ciblant les déubiquitinases, acteurs majeurs de ces processus biologiques. Ainsi les tests cellulaires permettant de quantifier la translocation nucléaire de NF-κB sous induction au TNFα ou d'évaluer l'autophagie (
Figure 2) dans les cellules ont été utilisés dans des criblages de composés chimiques en vue d'identifier de nouveaux modulateurs chimiques (Projets en cours). Ces mêmes essais ont été utilisés avec succès pour l'étude de de l'extinction génique par shRNA de certaines Ubiquitin Specific proteases, USP.
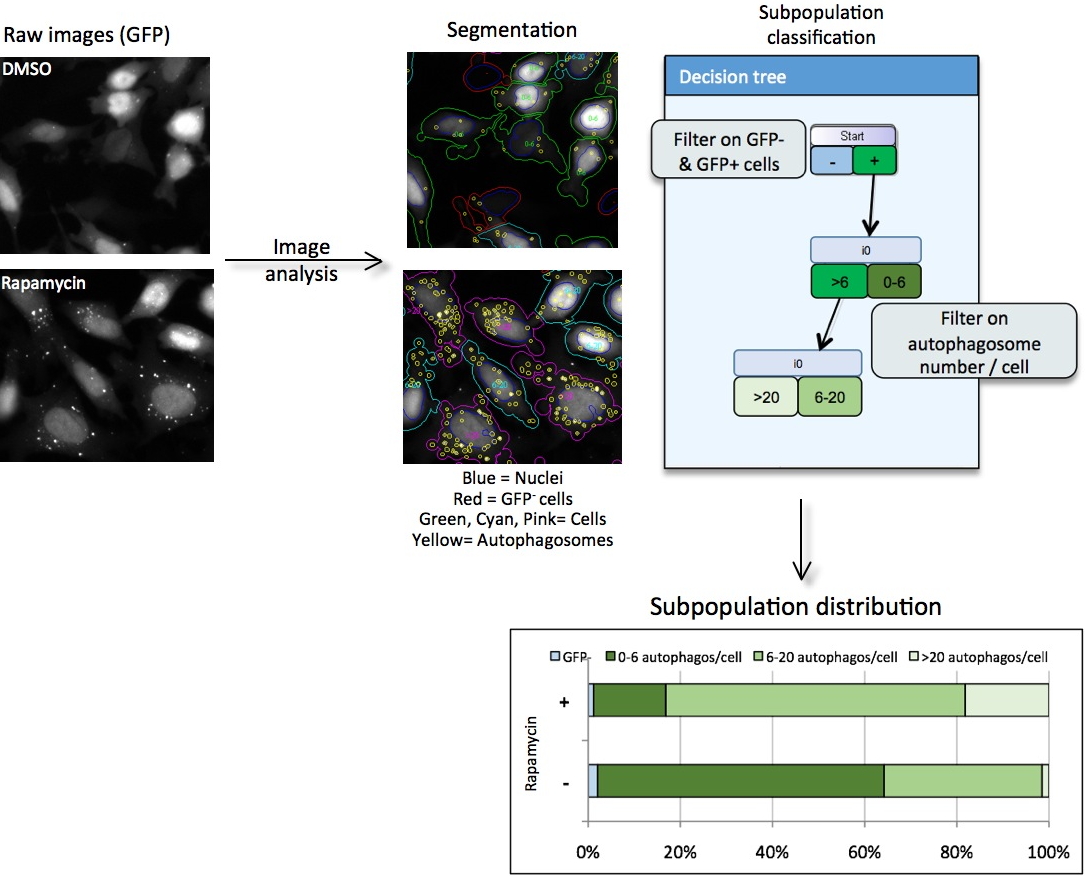 Figure 2 : Test cellulaire pour la quantification de l'autophagie.
Figure 2 : Test cellulaire pour la quantification de l'autophagie.
Des cellules HeLa exprimant la protéine LC3, marqueur spécifique des autophagosomes, couplée à la GFP ont été colorées avec du Hoechst pour la détection des noyaux. Les images des canaux de fluorescence Hoechst et GFP ont été acquises avec l’INCell Analyzer 1000 (objectif x20). Les images ont ensuite été analysées avec le logiciel INCell Investigator afin d’extraire des informations quantitatives et qualitatives comme le nombre de cellules, d'autophagosomes, l’intensité de fluorescence de chaque marquage, la forme ou encore l’aire des noyaux, des cellules et des autophagosomes. Depuis 2011, l'activité HCS de la plateforme a été ouverte aux utilisateurs de notre institut. Les modalités d'accès ainsi que le matériel disponible sont détaillés et expliqués dans une
charte s'adressant aux utilisateurs.
 Savoir faire et collaboration. Quelques exemples.
Savoir faire et collaboration. Quelques exemples.
• Tests cellulaires pour des applications HCS/HCA.