 Découverte et Evaluation de Biomarqueurs du Cancer de la Vessie
Découverte et Evaluation de Biomarqueurs du Cancer de la Vessie (DECANBIO)
 Coordinateur du projet
Coordinateur du projet : Jérôme Garin (EDyP)
Responsable EDyP : Christophe Masselon (EDyP)
Financement : Projet Européen FP7 (HEALTH) |
Le projet DECanBio conçoit et implémente une stratégie innovante pour la découverte et la validation de biomarqueurs protéiques. Notre approche est basée sur l'utilisation de nouvelles méthodes avancées de spectrométrie de masse pour l'analyse quantitative de protéines. La problématique pilote choisie est la détection précoce du cancer de la vessie dans l'urine. Le consortium
DECanBio réunit 7 partenaires originaires de 5 pays Européens : deux instituts de recherche cliniques, un établissement public de santé, trois laboratoires d'analyse protéomique et deux partenaires industriels. Le projet se subdivise en trois phases : La première phase a pour but la découverte de candidats biomarqueurs par de multiples approches méthodologiques (et notamment la
méthode AMT) dans une large cohorte muti-centrique. Lors de la seconde phase, ces candidats sont qualifiés par une méthode de protéomique ciblée (
SRM). La dernière phase du projet consiste en une vérification des candidats qualifiés dans une seconde cohorte, plus large et contenant des pathologies pouvant être confondues avec le cancer de la vessie.
Publications :
Makridakis M and Vlahou A Secretome proteomics for discovery of cancer biomarkers.
Journal of Proteomics, 2010
Selevsek N, Matondo M, Sanchez Carbayo M, Aebersold R and Domon B
Systematic quantification of peptides/proteins in urine using selected reaction monitoring.
Proteomics, 2011
Court M, Selevsek N, Matondo M, Allory Y, Garin J, Masselon CD and Domon B Towards a standardized urine proteome analysis methodology.
Proteomics, 2011
 Développement d'une approche intégrative d'identification de marqueurs impliqués dans la pathogenèse des lymphomes
Développement d'une approche intégrative d'identification de marqueurs impliqués dans la pathogenèse des lymphomes (CT_LYMPH)
 Coordinateur du projet
Coordinateur du projet : Saadi Khochbin et Mary Callanan (Institut Albert Bonniot, Grenoble)
Responsable interne : Anouk Emadali
Financement : INCA/DHOS - Recherche translationnelle |
Le projet est développé en étroite collaboration avec les équipes "
Epigénétique et Signalisation Cellulaire" (Saadi Khochbin) et "
Voies oncogéniques des hémopathies malignes" (Mary Callanan) de l'Institut Albert Bonniot à Grenoble
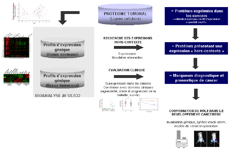
Dans la plupart des cancers, la transformation des cellules en cellules malignes résulte d'altérations génétiques ou épigénétiques qui peuvent se traduire
par l'expression aberrante de gènes spécifiques d'un tissu ou d'un type cellulaire particulier. Par exemple, certains gènes normalement exprimés exclusivement dans les testicules ont été trouvés comme étant sporadiquement dé-réprimés dans certains cancers somatiques. Le produit de ces gènes a été nommé CT pour
Cancer Testis. Pour un cancer donné, ces expressions illégitimes ont donc un
fort potentiel à représenter des biomarqueurs (Wang
et al.
2011). Une nouvelle stratégie d'identification de marqueurs de cancer centrée sur ces concepts a été développée dans le cadre de ce projet.
Dans cette approche originale, il ne s'agit donc pas de mettre en œuvre une analyse protéomique comparative entre des échantillons sains et cancéreux, mais de réaliser une simple
analyse protéomique d'échantillons pathologiques (ici des lignées cellulaires de lymphome) et d'utiliser des
données transcriptomiques afin
1) d'identifier des candidats marqueurs présentant un
profil d'expressions tissulaire « hors contexte » et
2) de
valider leur potentiel en utilisant les données cliniques (agressivité, stade et progression de la maladie, survie) associées à certaines études transcriptomiques publiques.
Ce projet a permis l'identification de plusieurs marqueurs potentiels. Parmi ces candidats, nous nous sommes concentrés, dans la deuxième phase de ce projet, sur une protéine présentant un profil d'expression « hors contexte », pour laquelle nous avons pu confirmer qu'elle était
surexprimée de façon aberrante dans les cancers lymphoïdes agressifs, mais également montrer qu'elle est associée à un
mauvais pronostic dans ces pathologies. En utilisant des approches d'
ARN interférant pour inactiver l'expression de cette protéine
in cellulo et
in vivo (modèle de xénotransplantation), nous avons pu valider son
rôle dans le développement tumoral et démontrer qu'elle était aussi impliquée dans la
réponse à l'immunothérapie anti-CD20 qui constitue à l'heure actuelle le traitement de référence des lymphomes. Ce marqueur diagnostique et pronostique peut donc également représenter une
cible thérapeutique pour une meilleure prise en charge clinique ce des cancers. Ces travaux ont donné lieu à un dépôt de
brevet européen et un manuscrit est en cours de soumission.
C'est dans le cadre de ce projet qu'a également été
mise en place l'approche SILAC et plus particulièrement l'
implémentation du logiciel MaxQuant (développé par le groupe de Matthias Mann au Max Planck Institute, Munich) au laboratoire pour étudier la réponse de lignées de lymphomes aux inhibiteurs d'histones désacétylases (Emadali
et al.
2009).
Publications :
Wang J, Emadali A, Le Bescont A, Callanan M, Rousseaux S and Khochbin S Induced malignant genome reprogramming in somatic cells by testis-specific factors.
Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms, 2011
Emadali A and Gallagher-Gambarelli M Quantitative proteomics by SILAC: Practicalities and perspectives for an evolving approach.
Medecine Sciences (Paris), 2009
Brevet :
Brevet européen : EP 11196053.0