Un des défis majeurs actuels dans le domaine de la santé consiste à découvrir de nouveaux biomarqueurs de pathologies ; le terme "biomarqueur de pathologie" pouvant être défini comme un paramètre biologique mesurable, indicateur d’un état pathologique. Les biomarqueurs de pathologie sont généralement détectés dans les fluides biologiques (sang, urine). La mesure précise de leur abondance permet de détecter de façon précoce certaines maladies (cancers par exemple), ou de suivre l'évolution de ces maladies suite à un traitement.
Aujourd'hui, les analyses protéomiques permettent d'analyser de manière fine le protéome (ensemble des protéines) d'échantillons cliniques issus d'un grand nombre de patients, et de comparer ce protéome à celui d'échantillons sains. À ce titre, de réels espoirs sont fondés sur la mise en œuvre d'analyses protéomiques pour découvrir des protéines qui seraient de nouveaux biomarqueurs de pathologies. Mais traduire des découvertes récentes en "outils" utilisables au quotidien en milieu hospitalier s'avère être un cheminement particulièrement long, difficile et coûteux. Ainsi, malgré des efforts intenses et des investissements lourds réalisés dans le domaine des approches "omiques" (protéomique, transcriptomique), le nombre de tests basés sur l'utilisation de nouveaux biomarqueurs de pathologies stagne.
Comment expliquer ce constat ?
En fait, quelle que soit la pathologie considérée, les approches à grande échelle menées dans le cadre d'études cliniques conduisent à l'identification de centaines de candidats biomarqueurs. Parmi ceux-ci, la plupart ne sont pas utilisables en médecine dans la mesure ou ils ne résultent pas directement du développement de la maladie mais correspondent plutôt à des perturbations biologiques secondaires. Beaucoup, par exemple, résultent de l'état inflammatoire des patients et sont donc communs à de nombreuses pathologies. Découvrir de nouveaux biomarqueurs spécifiques d'une pathologie par une approche "omique" impose donc d'être capable d'évaluer la pertinence (spécificité, sensibilité) de centaines de candidats biomarqueurs protéiques vis à vis de la maladie étudiée. Cette évaluation passe par la mesure précise de l'abondance de chacune de ces protéines dans les échantillons biologiques issus de grandes cohortes de malades, ces cohortes étant spécialement constituées dans le but de mener à bien des études statistiques pertinentes.
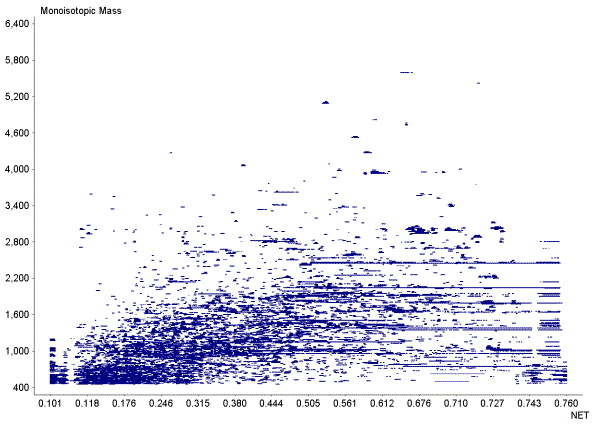 Carte protéomique de référence du protéome urinaire. Chaque point de la "carte" représente un peptide parfaitement caractérisé par son temps de rétention relatif en chromatographie de phase inverse (abscisse) d'une part, et la mesure précise de sa masse (ordonnée) d'autre part.
Carte protéomique de référence du protéome urinaire. Chaque point de la "carte" représente un peptide parfaitement caractérisé par son temps de rétention relatif en chromatographie de phase inverse (abscisse) d'une part, et la mesure précise de sa masse (ordonnée) d'autre part.
Classiquement, la mesure de l'abondance d'une protéine nécessite la production d'un anticorps qui reconnaît spécifiquement cette protéine, puis la mise au point d'un test de dosage qui utilise l'anticorps (test ELISA). Mais, pour des raisons de délai et de coût, il n'est pas envisageable de développer des centaines de tests ELISA pour évaluer la totalité des candidats biomarqueurs qui résultent d'une approche clinique de type "omique". Il semble donc que nous nous trouvions face à un mur ! Heureusement, une méthode de spectrométrie de masse ciblée (dite MRM, pour "Multiple Monitoring Reaction"), appliquée à l'analyse des protéines présentes dans les fluides biologiques, semble capable de lever ce verrou technologique. Des travaux très récents montrent en effet que la MRM, couplée à une étape de chromatographie liquide en amont, permet de réaliser en une heure l'analyse quantitative simultanée de plusieurs centaines de peptides présents dans un fluide biologique.
Le projet DECanBio, financé dans le cadre de la thématique "Santé" du 7ème PCRD, est coordonné par EDyP (Étude de la Dynamique des Protéomes). DECanBio est un projet de recherche "translationnelle" qui regroupe quatre laboratoires académiques européens, deux grands centres hospitaliers, et deux entreprises du domaine des biotechnologies. Pendant quatre ans, ces partenaires vont collaborer afin de découvrir et évaluer de nouveaux candidats biomarqueurs du cancer de la vessie en analysant le protéome urinaire de cohortes de malades, ainsi que le transcriptome (ensemble des ARN messagers) de tumeurs de la vessie. Ces travaux feront appel à des développements méthodologiques récents réalisés au sein de EDyP dans le domaine de la spectrométrie de masse à transformée de Fourier, de la MRM, et du traitement informatique des données. À l'issue de ce travail, un nouveau test de dépistage du cancer de la vessie devrait être mis au point.
La "plate-forme" technologique mise en place dans le cadre du projet DECanBio est générique et donc extrêmement prometteuse. En effet, une fois validée, cette plate-forme pourra être utilisée pour la recherche de biomarqueurs de pathologies très diverses, dans le domaine de la cancérologie, mais également des maladies cardio-vasculaires ou infectieuses.