Les génomes eucaryotes contiennent une quantité impressionnante d’éléments mobiles capables de se répliquer et de s’insérer en divers endroits du génome. Certains d’entre eux jouent un rôle vital dans l’expression génique ou le développement embryonnaire. Cependant, la transposition de ces éléments mobiles -ou transposons- favorise l’apparition de délétions, de duplications et de mutations constituant une source majeure d’instabilité génomique. Si cette instabilité contribue à l’évolution des génomes et l’adaptation des êtres vivants à leur environnement, elle peut aussi provoquer des mutations extrêmement délétères pour l’organisme. De nombreuses pathologies, dont plusieurs formes de cancers, sont ainsi associées à l’insertion de ces éléments mobiles dans des gènes ou oncogènes modifiant leur niveau d’expression. La répression de la transposition évite l’envahissement des génomes eucaryotes ainsi que l’apparition de mutations transmissibles à la génération suivante ; elle est critique pour le développement de la lignée germinale et la fertilité des individus.
Des études réalisées chez la mouche drosophile ou la souris ont démontré que la répression des transposons repose sur la genèse de petits ARN de la famille des ARNpi (PIWI interacting RNAs) par clivage de longs ARN simples brins transcrits à partir de loci spécifiques du génome, de transposons ou de séquences 3’ non traduites d’ARN messagers. Un cycle d’amplification induit la genèse de nouveaux ARNpi qui, en se liant à des protéines "PIWI", agissent à plusieurs niveaux dans le cytoplasme et dans le noyau sur l’hétérochromatine, afin de réprimer la transposition et l’expression de ces éléments mobiles. Les mécanismes déployés, dits épigénétiques, car modulant l'expression du patrimoine génétique sans toucher à sa séquence, demeurent encore mystérieux par bien des aspects.
Fort de nombreuses années d’expertise sur le modèle drosophile, nous avons répondu à la sollicitation du Dr Ramesh Pillai de l’EMBL, pour confirmer
in vivo sur des drosophiles génétiquement modifiées, des hypothèses élaborées
in cellulo sur des cellules d’insectes. L’ensemble de ces travaux identifient des séquences génomiques
[1] et des partenaires protéiques
[2] responsables de la production d'ARNpi chez la drosophile ou la souris (voir figure et sa légende pour plus de détails ).
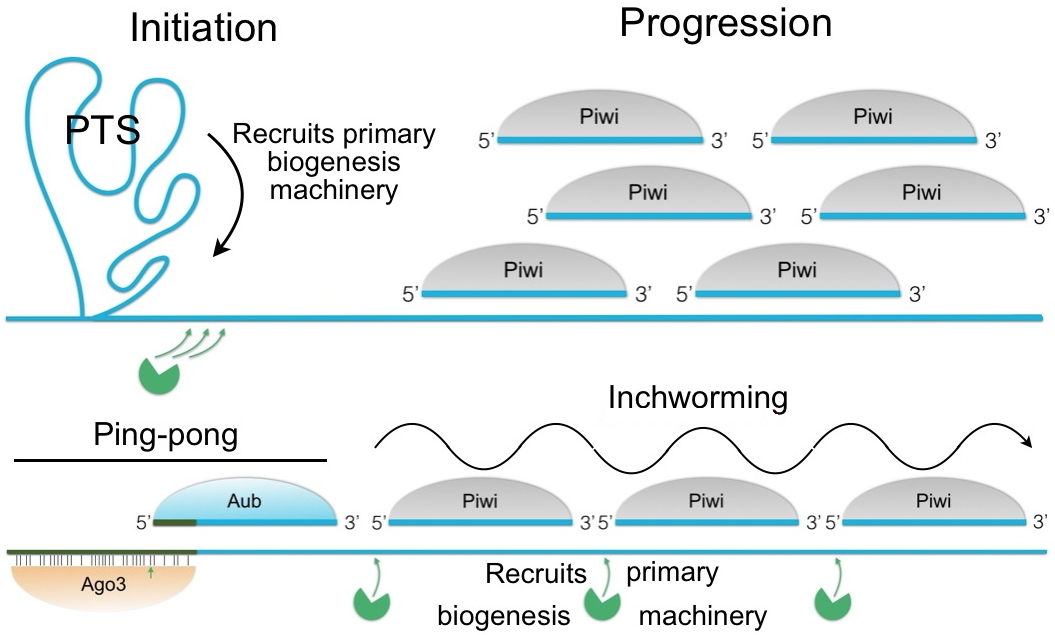
Biogenèse des ARNpi primaires.
Des gènes rapporteurs artificiels qui permettent de suivre et quantifier par séquençage exhaustif les ARNpi produits ont été introduits dans le génome de drosophile par transgenèse. Les résultats d’une première étude révèlent deux mécanismes distincts de recrutement des transcrits dans le processus primaire de clivage des transcrits. D’une part, un gène rapporteur contenant une séquence que les chercheurs de cette étude ont appelée PTS (piRNA-trigger sequence) en 5’ voit son transcrit clivé de façon directionnelle en piRNAs chevauchants représentant le premier pool de piRNAs (partie haute de la figure). D’autre part, le clivage du transcrit par le complexe PIWI cytosolique permet son entrée dans le processus de processing primaire qui résulte en la coupure du transcrit en ARNpi contigus et non-chevauchants en direction 3’. Ces derniers seront ensuite chargés dans le complexe PIWI nucléaire assurant le lien moléculaire entre répression cytoplasmique post-transcriptionnelle et répression de la transcription des loci correspondants dans le noyau.