Le projet CAPSI (Chaîne d’Analyse Protéomique multifactorielle haute Sensibilité à composants Intégrés) a pour objectif la détection et la quantification multiplexe de biomarqueurs protéiques dans des échantillons sanguins à l’aide d’une chaîne d’analyse protéomique miniaturisée. L’objectif visé à long terme est le développement d’outils diagnostiques innovants, de type "bornes de santé", permettant des analyses biomédicales plus rapides et plus fiables au sein des établissements de santé.
Des chercheurs de notre laboratoire sont spécialisés en analyse protéomique (analyse des protéines présentes dans des échantillons biologiques complexes). Ils ont développé et optimisé une chaîne d’analyse permettant la détection et la quantification exacte de biomarqueurs protéiques dans des échantillons sanguins. Cette chaîne est constituée d’une étape de digestion trypsique des protéines, suivie d’une analyse couplant chromatographie liquide (LC) et spectrométrie de masse (MS). La quantification exacte des biomarqueurs est obtenue grâce à la méthode PSAQ™ (Protein Standard Absolute Quantification) basée sur l’addition dans l’échantillon biologique de standards internes constitués par des biomarqueurs protéiques marqués à l’aide d’isotopes stables.
Le projet CAPSI réunit les expertises de notre laboratoire et du Leti/DTBS en vue du développement d’une chaîne d’analyse protéomique miniaturisée. Ce projet s’appuie notamment sur l’expertise du Leti/LCIV pour le développement de composants microfluidiques, du Leti/LFCM pour la fonctionnalisation de ces composants et sur l’évaluation et l’optimisation des chaînes d’analyse et du Leti/LE2S sur les approches de problèmes inverses pour analyser les mesures. La chaîne miniaturisée est composée de différents modules (Figure) dont une puce de digestion des protéines, un module de chromatographie liquide et un "nez" intégré permettant l’ionisation des peptides avant analyse par spectrométrie de masse.
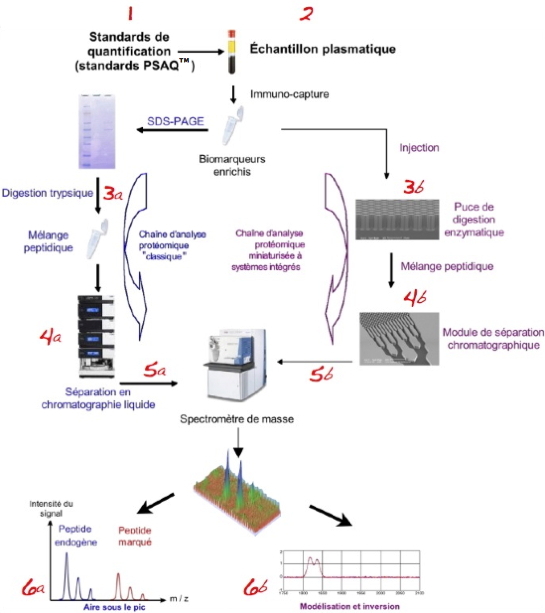
Représentation schématique de la détection et de la quantification multiplexe de biomarqueurs dans le sérum par analyse protéomique.
Les analyses sont réalisées en parallèle sur une chaîne d’analyse "classique" grâce à l'expertise du laboratoire EDyP (en bleu) et une chaîne miniaturisée à composants intégrés développée au Leti/DTBS (en violet).
1. Des standards de quantification (biomarqueurs incorporant des isotopes stables) sont ajoutés en quantité définie à l’échantillon.
2. Les biomarqueurs et les standards sont "extraits" de l’échantillon par immuno-capture.
Analyse protéomique "classique" (EDyP) :
3a. Les biomarqueurs et les standards sont digérés par la trypsine après une étape de SDS-PAGE.
4a. Les peptides trypsiques sont analysés par chromatographie liquide et spectrométrie de masse.
5a. La quantification est obtenue par comparaison des signaux MS des peptides issus des biomarqueurs et des peptides issus des standards.
6a. Identification des pics et l’extraction de leurs paramètres, puis la quantification des protéines cibles en faisant le rapport avec les paramètres de la protéine marquée.
Analyse sur chaîne à systèmes intégrés (Leti/DTBS) :
3b. Les biomarqueurs et les standards sont injectés dans une puce de digestion dont les "plots" sont greffés avec de la trypsine.
4b. Les peptides trypsiques sont injectés dans un module de séparation chromatographique doté d’un "nez" qui permet l’ionisation des peptides avant analyse par spectrométrie de masse.
5b. La quantification est obtenue par l’analyse des signaux MS des peptides issus des biomarqueurs en utilisant les signaux des standards pour contrôler les gains.
6b. Reconstruction directe des profils protéiques à partir des spectres par inversion d’un modèle de chaîne d’acquisition.
Les techniques de traitement de l’information développées dans l’équipe PROTIS dirigées par
Pierre Grangeat (DTBS/STD/LE2S) visent à optimiser l’analyse du signal MS de manière à réaliser une quantification des molécules cibles automatique, robuste vis-à-vis du bruit de mesure, et adaptative vis-à-vis des fluctuations sur la chaîne d’analyse (gain, temps de rétention,.. ). Afin de tester les performances de la chaîne d’analyse miniaturisée, des analyses quantitative des biomarqueurs choisis comme modèles sont réalisées en parallèle sur une chaîne d’analyse instrumentale "classique" utilisée en routine au sein de notre laboratoire, mais en tirant partie de la capacité de la méthode PSAQ™ à mesurer de manière exacte la quantité de protéines contenue dans un mélange complexe. Dans une telle configuration, il est possible de détecter et de quantifier des biomarqueurs dans le sérum à des niveaux pathologiques, mais également à des concentrations physiologiques de l’ordre du nanogramme par millilitre. Après avoir testé les composants de la chaîne d’analyse miniaturisée de manière indépendante, il reste désormais à les assembler et à les tester en série.
Le projet CAPSI est soutenu par le programme transverse du CEA « Technologies pour la Santé ».