La méthylation des protéines est une modification post-traductionnelle qui joue un rôle dans la régulation de presque tous les processus cellulaires de base. La grande majorité de nos connaissances sur la méthylation des protéines provient d’études menées sur les histones. Chez les plantes, au niveau du chloroplaste, seules quelques protéines (dont la Rubisco et les aldolases, des enzymes clef impliquées dans la fixation du CO2 au cours de la photosynthèse) ont été identifiées et leurs sites de méthylation déterminés.
Étant donné la position centrale occupée par les chloroplastes dans la physiologie des plantes, il est raisonnable de penser que la méthylation des protéines chloroplastiques puisse jouer un rôle dans de nombreux aspects du développement et de l’adaptation des plantes aux contraintes de l’environnement (sécheresse, stress métalliques etc…).
Des travaux précédemment réalisés par des chercheurs du laboratoire Physiologie Cellulaire & Végétale de notre institut ont montré que Rubisco et Aldolases sont triméthylées par une protéine méthyltransférase chloroplastique appelée LSMT, spécifique du monde végétal. Cependant, la modification de ces protéines par la LSMT est variable d’une espèce végétale à l’autre. Par exemple, les aldolases chloroplastiques sont naturellement méthylées chez le pois et Arabidopsis, alors que la Rubisco n’est méthylée que chez le pois. Dans ce contexte, ces chercheurs ont souhaité comprendre les mécanismes d’évolution moléculaire qui ont conduit à la variabilité de spécificité de substrat de la LSMT. Par des approches phylogénétiques et fonctionnelles (analyse biochimique de mutants et de chimères de LSMT), ils sont parvenus à identifier la triade His-Ala-Trp localisée dans la partie centrale de l’enzyme comme étant le motif clef conférant la capacité de méthyler la Rubisco (Figure) et de montrer que la fonction ancestrale de la LSMT était uniquement la méthylation des aldolases. La nature n’a ainsi eu besoin que de quelques mutations critiques pour aboutir à cette évolution (Figure) : une insertion essentielle (l’Alanine 259) accompagnée de deux substitutions (l’Histidine 252 en lieu et place d’une Tyrosine, et le
Tryptophane 265 en remplacement d’une Arginine).
Dans un contexte de recherche finalisée, le chloroplaste est le compartiment qui,
via la photosynthèse et l’assimilation du carbone, contrôle la biomasse végétale. L’analyse, actuellement en cours de lignées d’Arabidopsis produisant des LSMT avec des spécificités de substrat altérées, devrait apporter des éléments décisifs nécessaires à la compréhension du rôle de l’évolution de la méthylation des enzymes responsables de l’assimilation du carbone. De plus, ils faciliteront le transfert de nos connaissances entre espèces modèles et plantes de grandes cultures d’intérêt agronomique ou biotechnologique.
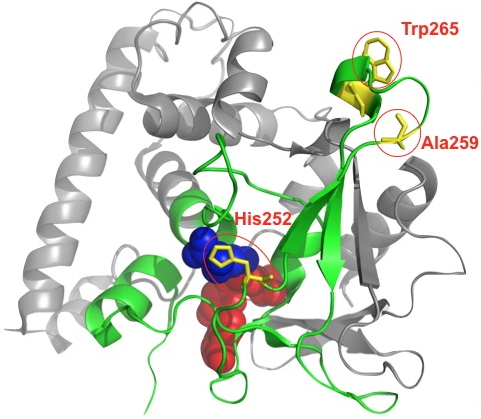
Analyse structurale de la LSMT de pois montrant en jaune les trois acides aminés essentiels pour la spécificité de substrat de l’enzyme vis-à-vis de la Rubisco : Alanine 259, Histidine 252 et Tryptophane 265.