Les petits ARN à interférence (siRNA) exogènes représentent un outil moléculaire puissant pour l'extinction ciblée de l'expression des gènes et largement utilisés pour l'étude fonctionnelle d'un gène ou le développement de nouvelles molécules d’intérêt thérapeutique. Bien que des progrès considérables aient été récemment accomplis dans la compréhension des mécanismes moléculaires sous-jacents de la voie à ARN interférence, les règles de conception (appelée également design) de siRNA potentiellement efficaces demeurent insatisfaisantes.
L’interférence ARN est un processus par lequel un ARN double-brin peut induire le clivage de l’ARNm dont il est complémentaire ou la répression de la traduction de cet ARNm en protéine, par encombrement stérique. Dans tous les cas, cette association conduit à l’extinction spécifique de l’expression d’un gène. Il est possible de mimer les effets de l’interférence ARN en utilisant des séquences d’ARN double brin synthétisés chimiquement de taille inférieure à 30 nucléotides (siRNA). Bien que toute molécule de siRNA puisse théoriquement inhiber l’expression génique de façon spécifique, le
design de ces séquences s’est rapidement avéré difficile. En effet, l’efficacité d’extinction se révèle hétérogène d’un siRNA à l’autre et d’autre part il faut éviter les effets hors-cibles dus à une hybridation partiellement spécifique des siRNA sur des ARNm non concernés.
En 2006, Yves Vandenbrouck de notre laboratoire, en collaboration avec l'école des Mines Paris avait conçu une méthode de prédiction de l'efficacité des siRNA. Est né
DSIR[1] (
Figure 1) (
Designer of Small Interfering RNA), un algorithme de design de séquences de siRNA. En 2007, DSIR a été classé par le NCBI (
National Center for Biotechnology Information) comme étant l’un des outils de conception de siRNA les plus performants. [Matveeva
et al.,
Nucleic Acids Research]

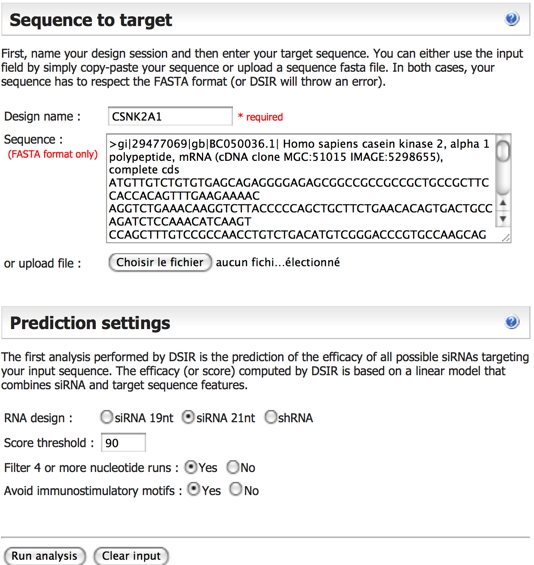
Figure 1 : DSIR (Designer of Small Interfering RNA). Page d’accueil. http://biodev.extra.cea.fr/DSIR
Dans le but d’évaluer les performances de DSIR, des chercheurs du laboratoire Biologie du Cancer et de l'Infection et de notre laboratoire viennent de réaliser [2] une étude grandeur nature mettant en œuvre un système standardisé de mesure de l’efficacité des siRNA. Avec le soutien de la Ligue Nationale contre le Cancer (programme Carte d’Identité des Tumeurs), ils ont utilisé DSIR pour concevoir 128 siRNA ciblant
10 gènes d’intérêt majeurs en cancérologie et dont l’activité à été systématiquement mesurée au niveau transcriptionnel et protéique (Figure 2).
Pour chaque gène, au moins 9 siRNA proposés par DSIR comme ayant une efficacité supérieure à 80% d’extinction ont été testés en les introduisant dans des cellules HeLa (cellules cancéreuses humaines). Cette étude révèle des performances de prédictions améliorées par rapport à l’état de l’art. De plus, des séquences conçues avec DSIR sont efficaces même à de très faibles concentrations (de l’ordre du nano molaire) permettant ainsi de minimiser les effets hors-cibles. Enfin l’analyse systématique des descripteurs (e.g. accessibilité à l’ARNm ciblé…) permet d’en infirmer ou d’en confirmer certains mais également d’identifier une relation positive entre la position dans le transcrit où le siRNA anti-sens vient s’hybrider et la distance à l’extrémité 5’.
Le
site Web DSIR intègre désormais ces améliorations, et met à disposition de la communauté scientifique la liste des siRNA testés qui pourront bénéficier aux équipes travaillant sur ces gènes d’intérêts en cancérologie ainsi qu’aux concepteurs de modèles de prédiction.
Les siRNA représentent un puissant outil d’investigation et leur utilisation s’est largement déployée dans les laboratoires de recherche pour des études de génomique fonctionnelle ou encore dans le but d’évaluer de nouvelles pistes thérapeutiques. À ce jour, le site web DSIR propose un service étendu de
design de siRNA. Il fait l’objet d’une utilisation intensive et est reconnu internationalement comme en témoigne la récente publication parue dans
Nature Methods issue de l’équipe de renom de Norbert Perrimon.
Gènes ciblés :
ERCC1, ERCC2, CSNK2A1, CSNK2A2, CSNK2B, HIF1A, BCL2L1, HDAC6, LIG1 et
PAR1