En amont de nombreuses pathologies il y a un moment où une cellule qui se divise donne naissance à une cellule fille différente de sa sœur et dont l’avenir sera cancéreux ou dégénératif. Si seulement on pouvait voir cet instant ? Très souvent en effet, cette cellule n’affichera aucun signe extérieur de ce changement. Comment alors tracer in vivo chaque étape de son devenir ? Comment également déterminer si ces changements intérieurs peuvent affecter tout un tissu ? C’est dans cette optique que Ruth Griffin de notre institut, a mis sur pied un projet international avec comme objectif de rendre disponible les outils nécessaires à l’analyse systématique des mécanismes moléculaires intervenant dans des maladies humaines. La phase I de ce projet est désormais achevée et vient de faire l’objet d’un article dans
Nature Methods [1].
Au cours d’une collaboration de quatre ans avec le Professeur N. Perrimon (Department of Genetics, Howard Hughes Medical Institute, Harvard Medical School, USA), Ruth Griffin a développé un nouvel outil génétique chez la drosophile qu’elle a baptisée le « Twin Spot Generator » ou TSG. L’originalité de cette approche réside dans sa dualité qui offre un moyen simple et efficace de tracer le destin de deux cellules filles issues d’une division cellulaire (Figure 1).
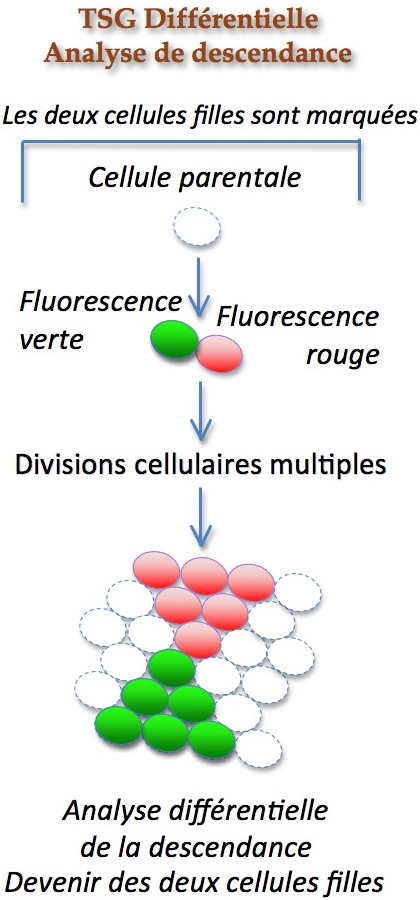
Figure 1. Analyse de descendance différentielle proposée par TSG.
TSG génère après la recombinaison mitotique deux « twin spots », ou cellules filles, rendues visibles et distinguables dans les tissus internes grâce à la fluorescence verte et rouge permettant une analyse différentielle de leur devenir. Précédemment une seule cellule pouvait être marquée [3].
La division cellulaire générera deux cellules filles, ou «
twin spots », marquées différentiellement juste après leur division. Le changement induit par TSG se situe au niveau de l’ADN, et de ce fait il est permanent et héréditaire. Pour la première fois, chez la drosophile, il devient possible de tracer
in vivo le destin des deux «
twin spots » dans le temps et à travers l’espace au cours d’analyses de descendance. Dans l’article de
Nature Methods[1], Griffin
et al. ont détecté une différence dans la cohésion cellulaire dans la descendance d’une des cellules filles (
Figure 2).
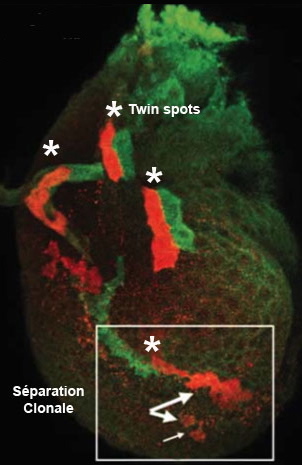
Figure 2. Perte de cohésion dans la descendance d’un « twin spot ».
Les astérisques marquent différent « twin spots » dans un tissu progéniteur de la patte de la mouche. Le rectangle entoure une région spécifique où les cellules de même origine (en rouge) se sont séparées par perte de cohésion. Ce comportement est spécifique à ce tissu.
De plus, ils ont réussi à visualiser les «
twin spots » dès la première division cellulaire. Ainsi, ils ont pu remonter dans le temps afin de définir le premier instant à partir duquel les deux cellules filles ont divergé (dans ce cas par leur forme :
Figure 3).
Au fur et à mesure du développement de TSG un autre projet d’envergure émergeait dans le laboratoire du Professeur Perrimon
[4]. Concrètement, il s’agissait de construire une collection de mouches transgéniques composées de 6 500 lignées indépendantes, possédant chacune une mutation induite par un ARNi (ARN interférence). Il est alors apparu à ces chercheurs qu’en combinant ces deux approches (biologie systémique et génétique), une synergie prendrait forme.
Dans cette
nouvelle approche appelée TSG-RNAi (Figure 4A), l’ARNi apporte l’aspect systématique en permettant d’introduire une mutation sous forme d’un ARNi à un seul des deux «
twin spots». Ainsi, il est possible de réaliser un criblage à haut débit pour identifier la fonction des gènes de la drosophile. Quant à l’outil génétique TSG, en présentant côte à côte les cellules sauvages et les cellules mutantes, il fournit un contrôle interne quasi parfait.
Les informations issues de ces cribles auront des implications dans la compréhension de l’évolution de certaines maladies humaines voire même dans leurs traitements par thérapie cellulaire. En effet, la drosophile est devenue un modèle central dans l’investigation biomédicale depuis que le séquençage des génomes entiers de l’Homme et de la mouche a montré que
plus de 60 % des gènes humains liés à des maladies sont similaires aux gènes de la mouche.
L’ensemble de ce travail s’est vu octroyer une bourse de quatre ans par le NIH (l’Institut National américain de la Santé) afin de démarrer un projet multidisciplinaire. Il est prévu, pour la première fois, que ces chercheurs, en concertation avec la communauté drosophile, cribleront
in vivo la fonction des gènes impliqués dans les maladies humaines. Il s’agit notamment de valider ceux qui avaient été identifiés par les criblages à haut débit dans les cellules en culture.
Grâce à sa "double vision", TSG peut ouvrir une toute nouvelle voie qui permettra de détecter une
régulation réciproque par échange d'informations entre cellules mutantes et normales (Figure 4B). Par exemple, il est possible qu'une mutation RNAi n'induisant aucun phénotype particulier dans les cellules qui la porte, puisse affecter la viabilité des cellules sœurs sauvages qui l'environnant. Un tel phénomène serait resté invisible dans les criblages précédents menés avec un seul marqueur.
* Chaque séquence d’ARN interférence a pour fonction d’inhiber de façon très spécifique son gène cible. Il devient ainsi possible d'observer ce qu'il se passe lorsque l'expression d'un gène particulier est fortement réduite.
* Dans ce travail Didier Grunwald de notre institut a réalisé l’acquisition et l’analyse des images confocales.