L’ingénierie métabolique vise à développer des micro-organismes particulièrement performants pour la production de molécules d’intérêt. Afin d’optimiser cette production, il est nécessaire d’avoir recours à des modèles de prédiction des flux métaboliques. Jusqu’à ce jour, faute de disposer d’une méthode permettant d’avoir accès aux quantités exactes d’enzymes présentes dans les cellules, les modèles développés n’intégraient pas ces données.
Les travaux réalisés dans notre laboratoire ont permis d’appliquer une stratégie analytique utilisant la spectrométrie de masse (technologie PSAQ™) à la quantification exacte et multiplexe de 22 enzymes intervenant dans le métabolisme central de la bactérie
Escherichia coli. Cette stratégie à permis de comparer l’abondance de ces enzymes entre une souche de référence d’Escherichia coli et deux souches modifiées mutées qui produisent un pouvoir réducteur élevé (quantités importantes de NADPH). Ces enzymes font partie de la voie de synthèse de l'acide tricarboxylique, des pentoses phosphates et de la glycolyse.
La combinaison de la connaissance de la quantité absolue de protéines et de données de fluxomique (étude des flux métaboliques) permet de définir le paramètre cinétique « F » qui correspond à l’activité enzymatique effective (
Figure) et de distinguer deux niveaux de régulation d’une réaction : soit au niveau de la quantité d’enzyme qui catalyse cette réaction, soit au niveau de l’activité enzymatique proprement dite. Cette information relative au niveau de régulation est capitale pour orienter la stratégie d’ingénierie métabolique puisque celle-ci cherchera à augmenter la quantité d’enzyme, ou à modifier l’activité de l’enzyme. De plus la connaissance de la quantité exacte d’enzyme présente dans la cellule est un élément clé pour déterminer la valeur Kcat/Km, souvent absente des bases de données d’enzymes, et nécessaire à toute modélisation.
Les résultats obtenus permettent de déterminer certains paramètres enzymatiques de voies métaboliques d'intérêt qui pourront être utilisés dans le cadre du développement d'outils prédictifs en biologie des systèmes et dans de nombreuses applications de biotechnologie.
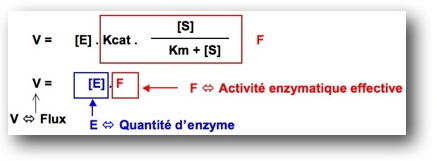
Equation de Michaelis-Menten et paramètres cinétiques associés.
V = flux. [E] = concentration de l’enzyme. Kcat = turnover. Km = constante d’affinité. [S] = concentration du substrat.
Ce travail est le fruit d’une collaboration entre l’équipe BGE/EDyP, l’entreprise MetabolicExplorer et l’équipe IBIS de l’Inria/Grenoble.
Cette stratégie est développée dans le cadre du projet RESET, Investissements d’Avenir Bio-Informatique (dont le porteur du projet est Hidde De Jong, Inria) visant à modéliser la machinerie d’expression génique des bactéries dans le but d’optimiser la production de composés d’intérêt.