La spectrométrie de masse s’est imposée comme une technique de référence en analyse protéomique, mais les données générées restent principalement qualitatives. Rendre les données de spectrométrie de masse quantitatives constitue donc un enjeu majeur.
La protéomique a vécu une véritable révolution technologique grâce aux avancées de la spectrométrie de masse (MS). Durant ces dernières années, un effort particulier de la communauté internationale a porté sur le développement de méthodes de
spectrométrie de masse quantitatives dédiées à la protéomique
[6].
Plusieurs méthodes génériques permettent actuellement une comparaison des concentrations en protéines présentes dans différents échantillons biologiques, mais ne fournissent pas d’information sur la quantité exacte (« absolue ») de chaque protéine présente. Pourtant, une
quantification exacte est requise dans des domaines clefs tels que l’analyse clinique, l’évaluation de candidats biomarqueurs, le contrôle qualité des médicaments ou des aliments, ou la surveillance sanitaire.
Suivant le principe de la dilution isotopique, une quantification exacte d’un composé peut être obtenue en spectrométrie de masse en ajoutant dans l’échantillon à analyser une quantité connue d’un étalon interne chimiquement identique au composé à doser, mais alourdi par l’incorporation d’isotopes stables. Ainsi, deux méthodes de quantification exacte utilisant des peptides marqués (13C et/ou
15N) comme étalons internes avaient été publiées (stratégies AQUA et QconCAT)
[1, 5] Ces méthodes reposaient toutes deux sur l’utilisation d’un peptide rapporteur par protéine à doser. Ce peptide rapporteur, marqué (13C et/ou
15N) est identique à un peptide trypsique identifié exclusivement dans la protéine à doser (peptide protéotypique).
Les toxines superantigéniques de
Staphylococcus aureus (entérotoxines et TSST-1) sont responsables de pathologies sévères chez l’homme [4] et représentent la seconde cause de toxi-infections alimentaires collectives après
Salmonella. Ces toxines peuvent contaminer la chaîne alimentaire et les réseaux d’eau potable. Leur détection et leur quantification par des approches immuno-chimiques reste problématique. En collaboration avec l’équipe du professeur F Vandenesch (Inserm U851, Lyon) et JA Hennekinne (AFSSA, Paris), nous avons donc testé une approche de spectrométrie de masse utilisant des étalons de type AQUA et QconCAT
[1, 5] pour la quantification des entérotoxines staphylo-cocciques. Les quantifications obtenues par cette approche se sont avérées inexactes. En effet, alors qu’elles permettent une quantification exacte (« absolue ») de la quantité du peptide ciblé dans l’hydrolysat trypsique de l’échantillon, elles ne prennent pas en compte les rendements de toutes les étapes biochimiques nécessaires à la préparation de l’échantillon.
Les auteurs ont donc développé la
méthode de quantification PSAQ™ qui utilise comme étalons internes des protéines recombinantes entières biochimiquement identiques aux protéines naturelles à doser. Ces étalons PSAQ™ sont alourdis par incorporation de [13C6,
15N2]L-lysine et [13C6,
15N4]L-arginine lors de leur biosynthèse en système « Cell-Free ». Les étalons internes PSAQ™, se comportant comme leurs «contre-parties» naturelles, peuvent être ajoutés préalablement à tout traitement de l’échantillon, ce qui permet d’éviter tout biais dû au processus analytique (Figure 1).
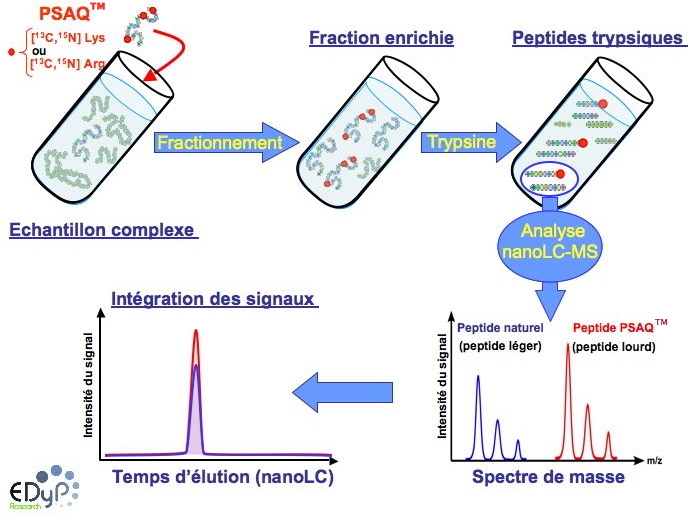
Figure 1 : Synopsis de la méthode PSAQ™
La méthode PSAQ™ repose sur la biosynthèse
in vitro d’étalons protéiques marqués isotopiquement. Ces étalons, biochimiquement équivalents aux protéines à doser, sont ajoutés à chaque échantillon préalablement à tout traitement. L’échantillon est ensuite fractionné, digéré à la trypsine puis analysé en nanoLC-MS. Chaque peptide issu d’un étalon PSAQ™ va ainsi produire un massif isotopique caractéristique décalé du peptide naturel à doser. L’intégration des signaux « léger » et « lourd » résultants nous permet de déterminer la concentration exacte du peptide cible.
La technique PSAQ™ est de fait la seule qui prenne en compte les rendements des étapes de pré-fractionnement, de décomplexification et de digestion trypsique. Une étude comparative a montré que seule la méthode PSAQ™ permettait une quantification exacte des toxines staphylococciques (Figure 2)
[2, 3].
Nous développons actuellement la méthode PSAQ™ dans plusieurs directions : i/ nous recherchons à repousser la limite de sensibilité de cette méthode sous le nanogramme de protéine cible par ml de matrice complexe (plasma, etc). ii / nous optimisons cette approche pour l’évaluation de biomarqueurs d’intérêt clinique. iii) enfin, nous mettons en place une plate-forme de production « haut-débit » et de calibration d’étalons PSAQ™ pour permettre des analyses multiplexes.
En conclusion, la méthode PSAQ™ s’avère beaucoup plus robuste et juste pour la quantification de protéines dans des échantillons biologiques complexes que les méthodes concurrentes développées précédemment. Sa justesse, sa précision, sa très grande spécificité et sa compatibilité avec des dosages multiplexes, font de cette méthode une alternative très intéressante à l’ELISA pour de nombreuses applications, dont l’évaluation à grande échelle de biomarqueurs dans les fluides biologiques tels que le sérum.
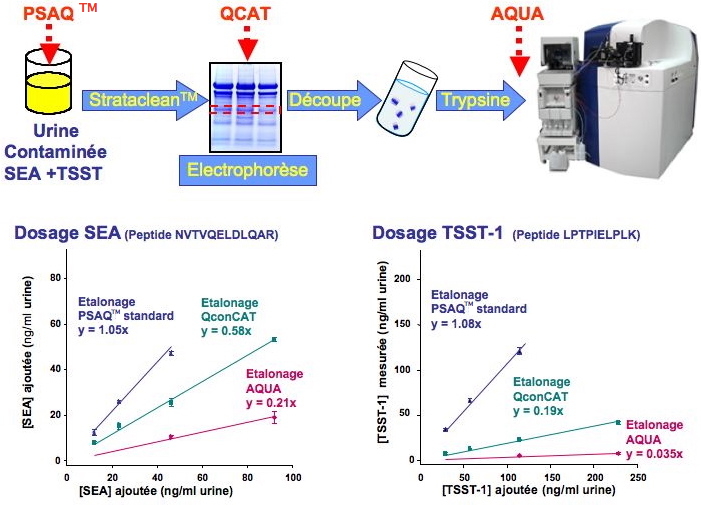
Figure 2 : Evaluation comparative des méthodes AQUA, QconCAT et PSAQ™ pour la quantification exacte des entérotoxines SEA et TSST.
Un échantillon d’urine est contaminé avec des quantités connues d’entérotoxines SEA et TSST (toxines naturelles). Chaque échantillon est divisé en trois, enrichi en toxine cible par concentration sur Strataclean™ et électrophorèse, et quantifié de manière comparative par les 3 méthodes. Les trois types d’étalon utilisés sont ajoutés comme indiqué par les flèches. Pour chaque toxine, la titration d’un même peptide protéotypique est représentée pour les trois méthodes (y = pente de la droite de titration).
Les prochaines expériences tendront à élucider la cascade de signalisation conduisant au découplage de l'annexine 2 du complexe jonctionnel ouvrant ainsi la voie vers la conception de nouveaux traitements anti-angiogéniques.